Construction of Recombinant Inbred Lines and Indentification of QTL for Grain Number Traits of Gaozhou Wild Rice
-
摘要:目的
野生稻(Oryza rufipogon)具有丰富的基因资源, 通过构建野生稻重组自交系并进行数量性状基因座(QTL)分析, 鉴定控制水稻穗粒数的QTL, 进而为水稻遗传改良提供分子基础和遗传资源。
方法通过高州野生稻和‘中花11’的杂交和多代自交, 构建了具有野生稻血缘的重组自交系群体。利用高密度基因芯片对群体进行基因型鉴定, 并基于分子标记分析构建了高密度遗传图谱。进一步对群体穗粒数表型进行2年考察, 结合表型数据和SNP标记信息, 利用QTL IciMapping软件进行了QTL分析。
结果成功构建了包含316个重组自交系的群体, 表型观察显示群体具有丰富的遗传多样性, 并在改良穗粒数上具有良好的应用潜力。检测到10个调控水稻穗粒数的QTL, 其中2022年检测到4个QTL, 2023年检测到6个QTL, 第11号染色体上的1个QTL表型贡献率最高、为13.78%。位于2号染色体上的QTL在两年中均可被稳定检测到。2年检测到的10个QTL中, 有9个加性效应为正值。
结论成功构建了高州野生稻重组自交系, 揭示出高州野生稻在改良水稻穗粒数中的应用潜力, 通过QTL分析揭示了控制水稻穗粒数的遗传基础。
Abstract:ObjectiveWild rice (Oryza rufipogon) possesses a wealth of genetic resources. The study aims to identify quantitative trait loci (QTLs) controlling the grain number per panicle in rice by constructing recombinant inbred lines (RILs) of wild rice and performing QTL analysis. This will provide a molecular basis and genetic resources for the genetic improvement of rice.
MethodA recombinant inbred line population with wild rice ancestry was developed through crosses between Gaozhou wild rice and 'Zhonghua 11', followed by multiple generations of selfing. High-density genotyping arrays were used to genotype the population, and a high-density genetic map was constructed based on molecular marker analysis. The phenotypic data for grain number per panicle were examined for two years. QTL analysis was conducted by using QTL IciMapping software with the combination of phenotypic data and SNP marker information.
ResultA population consisting of 316 recombinant inbred lines was successfully constructed. Phenotypic observations revealed rich genetic diversity within the population and good potential for application in improving the grain number per panicle. Ten QTLs regulating the grain number per panicle in rice were detected, among which four QTLs were detected in 2022 and six QTLs were detected in 2023. The phenotypic contribution of one QTL on chromosome 11 was the highest, accounting for 13.78%. The QTL located on chromosomes 2 was stably detected in two years. The additive effect was positive for 9 out of 10 QTLs detected at 2 years.
ConclusionThe recombinant inbred line of Gaozhou wild rice was successfully constructed, which revealed the application potential of Gaozhou wild rice in improving grain number per panicle of rice. QTL analysis revealed the genetic basis controlling the grain number per panicle in rice.
-
【研究意义】作为全球超过半数人口的主粮,水稻(Oryza sativa L.)产量与品质的不断提高对全球粮食安全的维护极为关键[1-4]。对我国这个世界最大的水稻生产和消费国而言,推进水稻遗传改良和产量提高已成为一项紧迫任务[5-6]。水稻的产量主要由有效穗数、穗粒数和粒重3个重要的构成因子决定。其中,每穗粒数是决定水稻产量的关键因素之一,与穗长、枝梗数和着粒密度关系密切,在高产品种的育种实践中,培育具有较多穗粒数的品种一直是育种家的主要育种目标之一。定位和克隆穗粒数调控基因对提高水稻产量具有重要的理论和应用价值。【前人研究进展】从农艺角度分析,穗粒数的形成受到小穗分化率和穗分化持续时间的影响。近年来,随着分子标记和全基因组测序等技术的飞速发展,研究人员已鉴定出多个与穗粒数相关的数量性状基因座(QTLs),并克隆了部分调控穗粒数的关键基因。例如,Gn1a和LOG基因通过影响细胞分裂素的合成与降解影响水稻穗粒数和产量[7-8];IPA1和NPT1在增加穗粒数的同时,减少分蘖数,增加粒重,显著提高水稻产量[9-10];调控水稻穗型的DEP1、LAEGE2通过影响分生组织活性进而调控水稻穗子大小和粒数[11-13];APO1和APO2参与水稻分生组织分化的起始时间影响小穗发育[14-15];Ghd7、Ghd2与穗分化相关且控制水稻抽穗期,同时调控水稻的抽穗期、株高和穗粒数[16-17]。水稻穗粒数调控基因的研究已取得一系列进展,这些有关穗发育调控基因的成功分离和克隆丰富了人们对水稻穗粒数性状调控分子机制的理解,为现代育种提供了有效的基因资源。【本研究切入点】水稻产量是一个极为复杂的农艺性状,同时受到多个性状因子的调控,这些性状表现出连续变异,一般认为受数量性状位点(QTL)、温度、光照等环境因素以及非生物胁迫的影响。水稻遗传机制复杂,已定位和报道的调控水稻穗部性状的QTL多达上千个,有大量控制水稻穗粒数的基因尚未被克隆,对水稻穗粒数形成的分子机制和遗传调控网络的理解仍不够深入。【拟解决的关键问题】本研究以高州野生稻(Oryza rufipogon)为材料,通过人工杂交和多代自交,构建了一组重组自交系(RILs)。这些重组自交系具有野生稻的遗传背景,但又能够在常规水稻栽培条件下进行有效的遗传分析。通过高密度芯片检测和分子标记分析,本研究构建了高密度的遗传连锁图谱,为QTL分析提供了精确的遗传框架。在此基础上,本研究针对穗粒数性状进行了QTL分析,旨在发掘控制穗粒数的关键QTL,为探索野生稻在水稻遗传改良中的应用潜力奠定基础。
1. 材料与方法
1.1 试验材料
本研究选取的供体材料为广东高州普通野生稻,与栽培稻同属AA基因组,可以通过传统方式与栽培稻进行杂交。材料取自华南农业大学野生稻种质资源圃,取样单株的资源编号为GZW-54。以粳稻‘中花11’为母本(45℃温汤处理母本5 min去雄)、高州野生稻为父本进行授粉,获得F1杂交种。然后通过单粒传法,经过多代自交,构建一套包含316个家系的重组自交系(RILs)群体。
1.2 田间种植与表型考察
水稻材料种植于广东省农业科学院水稻研究所大丰田间基地,采用宽窄行种植,株距13.3 cm,宽行行距23.1 cm、窄行行距16.5 cm,每个重组自交系种植2行。在2022年晚季和2023年晚季进行穗粒数表型的调查和基因型分析。每个家系选择表型一致的3个单株进行每穗粒数表型的考察,3个单株的算数平均值作为最终的每穗粒数表型数据。
1.3 基因型的获取
摘取水稻单株分蘖期的叶片,采用CTAB法提取植物DNA,利用武汉双绿源创芯科技研究院有限公司开发的高密度水稻基因芯片SLYm1R进行重组自交系基因系的鉴定。
1.4 分子标记与遗传图谱构建
在获得芯片信息后,对SNP标记进行数据过滤,筛选出高质量的SNP标记。通过QTL IciMapping软件构建高密度的遗传连锁图谱。
1.5 QTL分析
利用QTL IciMapping软件,采用完备区间作图法ICIM-ADD(Improved Composite Interval Mapping with Additional),结合表型数据和SNP标记信息,进行每穗粒数的QTL扫描,获得QTL的位置和贡献率、加性效应等数据。进一步利用R语言的ggplot包进行QTL图谱的绘制。
2. 结果与分析
2.1 重组自交系的构建
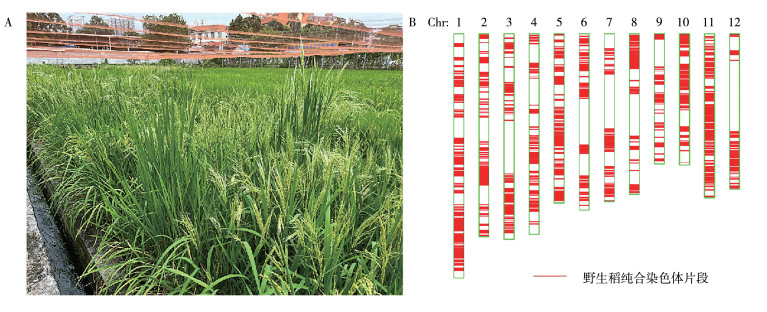
以高州野生稻为父本、粳稻‘中花11’为母本进行一次杂交获得F1种子,通过表型鉴定,选择与‘中花11’具有明显表型差异的单株作为真杂种进行收种,并种植F2群体。采用单粒传法进行连续自交,在F8代进行基因型的检测,并在F8代(2022年)和F9代(2023年)进行2次的表型考察。最终构建包含316个家系的一套重组自交系,这套具有高州野生稻血缘的重组自交系在田间表现出丰富的遗传多样性,并且基因组已基本纯合(图 1)。
2.2 重组自交系的育种价值评价
对重组自交系进行田间观察,发现与对照‘中花11’相比,多个重组自交系表现出穗长变长、穗粒数增多和产量增加等表型。对部分代表性增产重组自交系进行单株考种,结果(图 2)显示,穗长可增加30.0%~48.0%,平均穗粒数增加65.7%~89.0%,单株产量增加可达55.8%~90.4%。这表明来自高州野生稻的等位基因具有良好的育种应用价值,可以用来改良栽培稻品种,并且在改良水稻穗粒数上具有巨大的应用潜力。
![]() 图 2 重组自交系的植株表型与穗部表型A:对照‘中花11’;B~D:3个不同重组自交系;E~G:不同重组自交系的穗形态和穗粒数表型比较,左为‘中花11’;H~J:部分重组自交系穗长、平均穗粒数和单株产量的表型统计,数据展示的是平均值±标准差(双尾T检验,*为P < 0.05,**为P < 0.01)Figure 2. Plant phenotype and panicle phenotype of recombinant inbred linesA: 'Zhonghua 11'; B-D: Three different recombinant inbred lines; E-G: Comparison of panicle morphology and grain number of different recombinant inbred lines, and the left is 'Zhonghua 11'; H-J: Phenotypic statistics of panicle length, average grain number per panicle and grain yield per plant in partially recombined inbred lines; The data show the mean ± SD (two-tailed T test, *: P < 0.05, **: P<0.01)
图 2 重组自交系的植株表型与穗部表型A:对照‘中花11’;B~D:3个不同重组自交系;E~G:不同重组自交系的穗形态和穗粒数表型比较,左为‘中花11’;H~J:部分重组自交系穗长、平均穗粒数和单株产量的表型统计,数据展示的是平均值±标准差(双尾T检验,*为P < 0.05,**为P < 0.01)Figure 2. Plant phenotype and panicle phenotype of recombinant inbred linesA: 'Zhonghua 11'; B-D: Three different recombinant inbred lines; E-G: Comparison of panicle morphology and grain number of different recombinant inbred lines, and the left is 'Zhonghua 11'; H-J: Phenotypic statistics of panicle length, average grain number per panicle and grain yield per plant in partially recombined inbred lines; The data show the mean ± SD (two-tailed T test, *: P < 0.05, **: P<0.01)2.3 重组自交系SNP数据的获取
利用SLYm1R高密度水稻全基因组SNP芯片对重组自交系进行基因型的检测。SLYm1R芯片基于Illumina的芯片制造技术,包含31 753个SNP位点。以MSU7.0版本‘日本晴’为参考基因组,其中31 704个位点有坐标物理位置信息(图 3)。对获取的31 753个SNP标记进行数据过滤,利用QTL IciMapping软件的Bin识别,删除数据集中的冗余标记,对每条染色体的标记进行简化,得到7 775个高质量的SNP标记。进一步保留每条染色体的首尾标记,其余标记每隔3个取1个,最终得到2 591个SNP标记用于后续分析。
2.4 遗传图谱的构建
使用QTL IciMapping软件选择优化筛选的2 591个SNP标记构建遗传连锁图谱,选择仅基于锚定信息(Anchor only)进行分组,在所有标记正确分组后选择k-Optimality(k-最优)算法进行排序,并对已排序染色体进行精细调整,在所有染色体正确分组和排序之后绘制遗传图谱,如图 4所示。
2.5 水稻每穗粒数调控的QTL分析
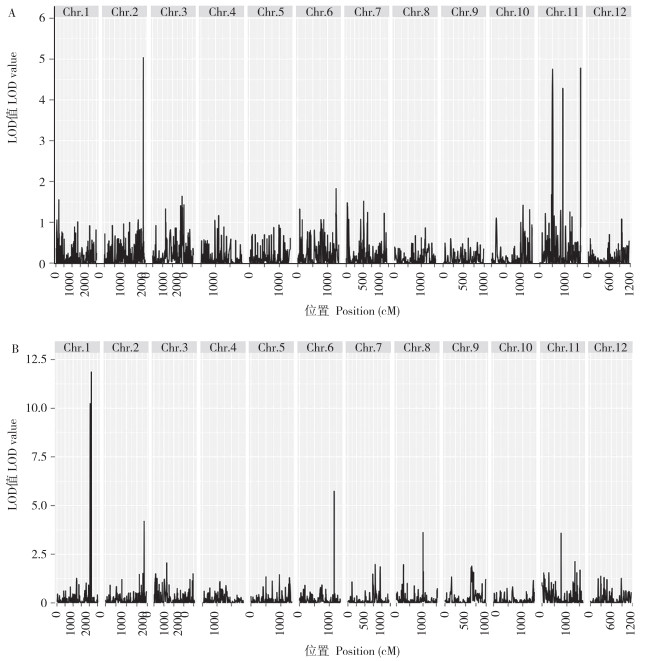
利用QTL IciMapping软件,采用完备区间作图法,对316个重组自交系的每穗粒数性状和SNP基因型数据进行QTL分析,LOD值设定为1 000次置换检验,检测步长为1 cM。根据计算得到的LOD值绘制QTL图谱(图 5)。进一步以LOD大于2.5作为阈值筛选,共检测到10个调控水稻每穗粒数的QTL(表 1),其中2022年检测到4个QTL,1个位于第2号染色体,3个位于第11号染色体,表型贡献率为5.02%~13.78%;2023年检测到6个QTL,位于水稻第1、2、6、8和11号染色体,表型贡献率为3.29%~8.74%。位于第2号染色体的qAGN-2在两年中均可被稳定检测到,LOD值在2022年和2023年分别为5.03和4.19,且定位位置较为接近,遗传效应稳定。值得注意的是,2年检测到的10个QTL中,有9个加性效应为正值,表明来自野生稻的等位基因可以增加每穗粒数表型,可以用于分子辅助育种、改良栽培稻的每穗粒数表型。
表 1 重组自交系穗粒数表型2年QTL定位结果Table 1. QTL mapping results for grain number per panicle of recombinant inbred lines in two years
3. 讨论
野生稻作为栽培稻的近缘种,是宝贵的基因资源库,尤其在抗病、抗虫、耐旱、耐寒等抗逆性状方面[18-24]。高州野生稻是广东境内最具代表性、分布面积最广的普通野生稻,可能是广东乃至我国的野生稻分化中心[25-27]。随着环境变化和人类活动的加剧,高州野生稻的自然生境受到严重威胁,其种质资源的保护和创新利用显得尤为重要。
野生稻基因库中存在着丰富的遗传多样性,这为水稻育种提供了宝贵的基因资源,同时也增加了基因挖掘和利用的难度,使其遭遇一系列挑战和技术难点。野生稻与栽培稻之间显著的遗传差异,往往导致两者在杂交过程中出现杂交不亲和现象,甚至产生不育的后代[28];许多具有潜在价值的产量提升和抗逆性基因与一些不利的农艺性状紧密连锁[29],难以通过常规的育种手段进行有效的分离和重组,使得优良基因的利用和推广受到限制。为克服这些障碍,育种工作者需要不断创新和优化育种技术,以便更有效地利用野生稻中的宝贵基因资源。
重组自交系在远缘杂交的基因资源利用中可以发挥重要作用[30-31]。通过构建野生稻与栽培稻的重组自交系,可以在高分辨率的遗传图谱上精细定位控制产量等性状的QTL,提高基因挖掘的精确性。通过杂交和随后的多代自交,重组自交系的创建过程中会增加基因重组机会,从而有可能将有利基因与不利基因分离,有助于打破连锁累赘。高代重组自交系具有固定的遗传背景,可以进行多年多点的表型调查,减少环境和遗传背景对表型评估的影响,使得基因效应更容易被检测和分析。
穗粒数是一个复杂的遗传性状,它受到少数主效数量性状基因座(QTL)和多个微效QTL的共同影响。在分子育种领域,表型贡献率超过10% 的主效QTL通常被认为具有重要的应用价值[32]。这些主效QTL的识别和利用,可以显著提升育种效率,加速优良性状的聚合和新品种的开发。在本研究的QTL分析中,我们在2年中共检测到10个与每穗粒数相关的QTL,这些QTL中有9个加性效应为正值,来自高州野生稻的基因可以增加每穗粒数,其中1个位于第11号染色体QTL的表型贡献率为13.78%。这一发现表明,高州野生稻中存在多个能够显著影响水稻的穗粒数性状,且在后续的分子辅助育种改良中具有良好的应用前景。来自中国农业大学的研究者利用元江野生稻鉴定出一个调控水稻穗粒数的基因GN2[33],与本研究2年中均可稳定检测到的qAGN-2,均位于2号染色体长臂末端相近的位置。值得注意的是,来自元江野生稻的等位基因表现为减少穗粒数,而来自高州野生稻的qAGN-2等位基因则可以增加穗粒数。这些发现揭示了不同野生稻种质资源中基因位点对穗粒数影响的差异性,qAGN-2与GN2之间的具体关系及其在不同种质资源中的作用机制,需要进一步深入研究来阐明。利用重组自交系进行高州野生稻基因资源的研究,可以有效发掘和转移高州野生稻中的优异等位基因到栽培稻中,提高水稻的穗粒数和产量,对于保障粮食安全和农业可持续发展具有重要意义。
4. 结论
本研究通过高州野生稻和‘中花11’的杂交和多代自交,构建了一套包含316个家系的重组自交系群体,利用高密度基因芯片,分两年对每穗粒数进行了QTL分析,共检测到10个调控水稻每穗粒数的QTL。其中,2022年检测到4个、2023年检测到6个,位于第2号染色体的qAGN-2在两年中均可被稳定检测到。2年检测到的10个QTL中,有9个加性效应为正值,表明来自野生稻的等位基因可以增加每穗粒数表型,揭示了高州野生稻在水稻遗传改良中的潜在应用价值。
【责任编辑 张辉玲】 -
图 2 重组自交系的植株表型与穗部表型
A:对照‘中花11’;B~D:3个不同重组自交系;E~G:不同重组自交系的穗形态和穗粒数表型比较,左为‘中花11’;H~J:部分重组自交系穗长、平均穗粒数和单株产量的表型统计,数据展示的是平均值±标准差(双尾T检验,*为P < 0.05,**为P < 0.01)
Figure 2. Plant phenotype and panicle phenotype of recombinant inbred lines
A: 'Zhonghua 11'; B-D: Three different recombinant inbred lines; E-G: Comparison of panicle morphology and grain number of different recombinant inbred lines, and the left is 'Zhonghua 11'; H-J: Phenotypic statistics of panicle length, average grain number per panicle and grain yield per plant in partially recombined inbred lines; The data show the mean ± SD (two-tailed T test, *: P < 0.05, **: P<0.01)
表 1 重组自交系穗粒数表型2年QTL定位结果
Table 1 QTL mapping results for grain number per panicle of recombinant inbred lines in two years

-
[1] HUANG X H, KURATA N, WEI X H, WANG Z X, WANG A, ZHAO Q, ZHAO Y, LIU K Y, LU H Y, LI W J, GUO Y L, LU Y Q, ZHOU C C, FAN D L, WENG Q J, ZHU C R, HUANG T, ZHANG L, WANG Y C, FENG L, FURUUMI H, KUBO T, MIYABAYASHI T, YUAN X P, XU Q, DONG G J, ZHAN Q L, LI C Y, FUJIYAMA A, TOYODA A, LU T T, FENG Q, QIAN Q, LI J Y, HAN B. A map of rice genome variation reveals the origin of cultivated rice[J]. Nature, 2012, 490(7421): 497-501. DOI: 10.1038/nature11532.
[2] BAILEY-SERRES J, PARKER J E, AINSWORTH E A, OLDROYD G, SCHROEDER J I. Genetic strategies for improving crop yields [J]. Nature, 2019, 575(7781): 109-118. DOI: 10.1038/s41586-019-1679-0.
[3] HICKEY L T, N H A, ROBINSON H, JACKSON S A, LEALBERTIOLI S, TESTER M, GAO C X, GODWIN I D, HAYES B J, WULFF B. Breeding crops to feed 10 billion[J]. Nature Biotechnology, 2019, 37(7): 744-754. DOI: 10.1038/s41587-019-0152-9.
[4] CHEN R Z, DENG Y W, DING Y L, GUO J X, QIU J, WANG B, WANG C S, XIE Y Y, ZHANG Z H, CHEN J X, CHEN L T, CHU C C, HE G C, HE Z H, HUANG X H, XING Y Z, YANG S H, XIE D X, LIU Y G, LI J Y. Rice functional genomics: Decades' efforts and roads ahead[J]. Science China Life Sciences, 2022, 65(1): 33-92. DOI: 10.1007/s11427-021-2024-0.
[5] 胡培松, 翟虎渠, 万建民. 中国水稻生产新特点与稻米品质改良[J]. 中国农业科技导报, 2002, 4(4): 33-39. HU P S, ZHAI H Q, WAN J M. New characteristics of rice production and quality improvement in China[J]. Review of China Agricultural Science and Technology, 2002, 4(4): 33-39.
[6] 郭韬, 余泓, 邱杰, 李家洋, 韩斌, 林鸿宣. 中国水稻遗传学研究进展与分子设计育种[J]. 中国科学: 生命科学, 2019, 49(10): 1185-1212. DOI: 10.1360/SSV-2019-2029. GUO T, YU H, QIU J, LI J Y, HAN B, LIN H X. Advances in rice genetics and breeding by molecular design in China[J]. Scientia Sinica Vitae, 2019, 49(10): 1185-1212. DOI: 10.1360/SSV-2019-2029.
[7] ASHIKARI M, SAKAKIBARA H, LIN S Y, YAMAMOTO T, TAKASHI T, NISHIMURA A, ANGELES E R, QIAN Q, KITANO H, MATSUOKA M. Cytokinin oxidase regulates rice grain production[J]. Science, 2005, 309(5735): 741-745. DOI: 10.1126/science.1113373.
[8] KURAKAWA T, UEDA N, MAEKAWA M, KOBAYASHI K, KOJIMA M, NAGATO Y, SAKAKIBARA H, KYOZUKA J. Direct control of shoot meristem activity by a cytokinin-activating enzyme[J]. Nature, 2007, 445(7128): 652-655. DOI: 10.1038/nature05504.
[9] WANG S S, WU K, QIAN Q, LIU Q, LI Q, PAN Y J, YE Y F, LIU X Y, WANG J, ZHANG J Q, LI S, WU Y J, FU X D. Non-canonical regulation of SPL transcription factors by a human OTUB1-like deubiquitinase defines a new plant type rice associated with higher grain yield[J]. Cell Research, 2017, 27(9): 1142-1156. DOI: 10.1038/cr.2017.98.
[10] JIAO Y Q, WANG Y H, XUE D W, WANG J, YAN M X, LIU G F, DONG G J, ZENG D L, LU Z F, ZHU X D, QIAN Q, LI J Y. Regulation of OsSPL14 by OsmiR156 defines ideal plant architecture in rice[J]. Nature Genetics, 2010, 42(6): 541-544. DOI: 10.1038/ng.591.
[11] HUANG L J, HUA K, XU R, ZENG D L, WANG R C, DONG G J, ZHANG G Z, LU X L, FANG N, WANG D K, DUAN P G, ZHANG B L, LIU Z P, LI N, LUO Y H, QIAN Q, YAO S G, LI Y H. The LARGE2-APO1/APO2 regulatory module controls panicle size and grain number in rice[J]. Plant Cell, 2021, 33(4): 1212-1228. DOI: 10.1093/plcell/koab041.
[12] SUN H Y, QIAN Q, WU K, LUO J J, WANG S S, ZHANG C W, MA Y F, LIU Q, HUANG X Z, YUAN Q B, HAN R X, ZHAO M, DONG G J, GUO L B, ZHU X D, GOU Z H, WANG W, WU Y J, LIN H X, FU X D. Heterotrimeric G proteins regulate nitrogen-use efficiency in rice[J]. Nature Genetics, 2014, 46(6): 652-656. DOI: 10.1038/ng.2958.
[13] HUANG X Z, QIAN Q, LIU Z B, SUN H Y, HE S Y, LUO D, XIA G M, CHU C C, LI J Y, FU X D. Natural variation at the DEP1 locus enhances grain yield in rice[J]. Nature Genetics, 2009, 41(4): 494-497. DOI: 10.1038/ng.352.
[14] KAWAKATSU K I, MAEKAWA M, IZAWA T, ITOH J I, NAGATO Y. ABERRANT PANICLE ORGANIZATION 2/RFL, the rice ortholog of Arabidopsis LEAFY, suppresses the transition from inflorescence meristem to floral meristem through interaction with APO1[J]. The Plant Journal, 2015, 69(1): 168-180. DOI: 10.1111/j.1365-313X.2011.04781.x.
[15] IKEDA-KAWAKATSU K, YASUNO N, OIKAWA T, IIDA S, NAGATO Y, MAEKAWA M, KYOZUKA J. Expression level of ABERR ANT PANICLE ORGANIZATION1 determines rice inflorescence form through control of cell proliferation in the meristem [J]. Plant Physiology, 2009, 150(2): 736-747. DOI: 10.1104/pp.109.136739.
[16] LIU J H, SHEN J Q, XU Y, LI X H, XIAO J H, XIONG L Z. Ghd2, a CONSTANS-like gene, confers drought sensitivity through regulation of senescence in rice[J]. Journal of Experimental Botany, 2016, 67(19): 5785-5798. DOI: 10.1093/jxb/erw344.
[17] WENG X Y, WANG L, WANG J, HU Y, DU H, XU C G, XING Y Z, LI X H, XIAO J H, ZHANG Q F. Grain number, plant height, and heading date7 is a central regulator of growth, development, and stress response[J]. Plant Physiology, 2014, 164(2): 735-747. DOI: 10.1104/pp.113.231308.
[18] 翟李楠, 唐清杰, 周世圳, 周帮纪, 云勇, 王惠艰, 韩义胜, 邢福能, 严小微. 海南普通野生稻稻瘟病抗性鉴定与评价[J]. 植物遗传资源学报, 2024, 25(1): 39-51. DOI: 10.13430/j.cnki.jpgr.20230523003. ZHAI L N, TANG Q J, ZHOU S Z, ZHOU B J, YUN Y, WANG H J, HAN Y S, XING F N, YAN X W. Identification and evaluation of resistance to rice blast in Oryza rufipogon Griff. in Hainan Province [J]. Journal of Plant Genetic Resources, 2024, 25(1): 39-51. DOI: 10.13430/j.cnki.jpgr.20230523003.
[19] YANG Z Y, XU Z J, YANG Q W, QIAO W H. Conservation and utilization of genetic resources of wild rice in China[J]. Rice Science, 2022, 29(3): 216-224. DOI: 10.1016/j.rsci.2021.08.003.
[20] ZHAO Q, FENG Q, LU H Y, LI Y, WANG A, TIAN Q L, ZHAN Q L, LU Y Q, ZHANG L, HUANG T, WANG Y C, FAN D L, ZHAO Y, WANG Z Q, ZHOU C C, CHEN J Y, ZHU C R, LI W J, WENG Q J, XU Q, WANG Z X, WEI X H, HAN B, HUANG X H. Pan-genome analysis highlights the extent of genomic variation in cultivated and wild rice[J]. Nature Genetics, 2018, 50(2): 278-284. DOI: 10.1038/s41588-018-0041-z.
[21] WING R A, PURUGGANAN M D, ZHANG Q F. The rice genome revolution: From an ancient grain to Green Super Rice[J]. Nature Reviews Genetics, 2018, 19(8): 505-517. DOI: 10.1038/s41576-018-0024-z.
[22] STEIN J C, YU Y, COPETTI D, ZWICKL D J, ZHANG L, ZHANG C J, CHOUGULE K, GAO D Y, IWATA A, GOICOECHEA J L, WEI S, WANG J, LIAO Y, WANG M H, JACQUEMIN J, BECKER C, KUDRNA D, ZHANG J W, LONDONO C, SONG X, LEE S, SANCHEZ P, ZUCCOLO A, AMMIRAJU J, TALAG J, DANOWITZ A, RIVERA L F, GSCHWEND A R, NOUTSOS C, WU C C, KAO S M, ZENG J W, WEI F J, ZHAO Q, FENG Q, BAIDOURI M E, CARPENTIER M C, LASSERRE E, COOKE R, FARIAS D R, MAIA L C, DOS SANTOS R S, NYBERG K G, MCNALLY K L, MAULEON R, ALEXANDROV N, SCHMUTZ J, FLOWERS D, FAN C Z, WEIGEL D, JENA K K, WICKER T, CHEN M S, HAN B, HENRY R, HSING Y C, KURATA N, OLIVEIRA A C, PANAUD O, JACKSON S A, MACHADO C A, SANDERSON M J, LONG M Y, WARE D, WING R A. Genomes of 13 domesticated and wild rice relatives highlight genetic conservation, turnover and innovation across the genus Oryza [J]. Nature Genetics, 2018, 50(2): 285-296. DOI: 10.1038/s41588-018-0040-0.
[23] LI W, LI K, HUANG Y, SHI C, HU W S, ZHANG Y, ZHANG Q J, XIA E H, HUTANG G R, ZHU X G, LIU Y L, LIU Y, TONG Y, ZHU T, HUANG H, ZHANG D, ZHAO Y, JIANG W K, YUAN J, NIU Y C, GAO C W, GAO L Z. SMRT sequencing of the Oryza rufipogon genome reveals the genomic basis of rice adaptation[J]. Communications Biology, 2020, 3(1): 167. DOI: 10.1038/s42003-020-0890-8.
[24] 廖冰, 黄秀艳, 陈科, 傅雪琳, 何平. 野生稻单片段代换系苗期耐旱性评价及QTL鉴定[J]. 广东农业科学, 2024, 51(3): 70-80. DOI: 10.16768/j.issn.1004-874X.2024.03.007. LIAO B, HUANG X Y, CHEN K, FU X L, HE P. Evaluation of drought tolerance and identification of QTL in single-segment substitution lines of wild rice at seedling stage[J]. Guangdong Agricultural Sciences, 2024, 51(3): 70-80. DOI: 10.16768/j.issn.1004-874X.2024.03.007.
[25] 陈志雄, 王兰, 吴锦文, 刘向东. 丁氏稻种资源有利基因挖掘与创新研究进展[J]. 华南农业大学学报, 2023, 44(5): 649-658. DOI: 10.7671/j.issn.1001-411X.202307016. CHEN Z X, WANG L, WU J W, LIU X D. Research progress in favorable gene mining and innovation of Ting's rice germplasm[J]. Journal of South China Agricultural University, 2023, 44(5): 649-658. DOI: 10.7671/j.issn.1001-411X.202307016.
[26] 李晨, 潘大建, 毛兴学, 涂从勇, 周汉钦, 范芝兰, 李晓方. 用SSR标记分析高州野生稻的遗传多样性[J]. 科学通报, 2006(5): 551-558. LI C, PAN D J, MAO X X, TU C Y, ZHOU H Q, FAN Z L, LI X F. Analyzing the genetic diversity of Gaozhou wild rice using SSR markers[J]. Science Bulletin, 2006(5): 551-558.
[27] 杨庆文, 张万霞, 时津霞, 任军方, 苗晗. 广东高州普通野生稻(Oryza rufipogon Griff. ) 的遗传多样性和居群遗传分化研究[J]. 植物遗传资源学报, 2004(4): 315-319. DOI: 10.13430/j.cnki.jpgr.2004.04.002. YANG Q W, ZHANG W X, SHI J X, REN J F, MIAO H. Genetic diversity and differentiation of Oryza rufipogon populations in Gaozhou[J]. Journal of Plant Genetic Resources, 2004(4): 315-319. DOI: 10.13430/j.cnki.jpgr.2004.04.002.
[28] 李莉萍, 应东山, 张如莲. 野生稻优良基因资源的研究与应用进展[J]. 热带农业科学, 2014, 34(1): 34-41. LI L P, YING D S, ZHANG R L. Research and application progress of valuable genes of wild rice[J]. Chinese Journal of Tropical Agriculture, 2014, 34(1): 34-41.
[29] 邓启云, 袁隆平, 梁凤山, 李继明, 李新奇, 王乐光, 王斌. 野生稻高产基因及其分子标记辅助育种研究[J]. 杂交水稻, 2004, 19(1): 6-10. DOI: 10.16267/j.cnki.1005-3956.2004.01.002. DENG Q Y, YUAN L P, LIANG F S, LI J M, LI X Q, WANG L G, WANG B. Studies on yield-enhancing genes from wild rice and their marker-assisted selection in hybrid rice[J]. Hybrid Rice, 2004, 19(1): 6-10. DOI: 10.16267/j.cnki.1005-3956.2004.01.002.
[30] 彭强, 徐海峰, 宫彦龙, 吴娴, 吴朝昕, 吴健强, 朱速松. 基于高密度遗传图谱的水稻糙米籽粒大小QTL定位[J]. 广东农业科学, 2023, 50(12): 96-103. DOI: 10.16768/j.issn.1004-874X.2023.12.009. PENG Q, XU H F, GONG Y L, WU X, WU C X, WU J Q, ZHU S S. Mapping of QTLs for rice grain size based on high-density genetic map [J]. Guangdong Agricultural Sciences, 2023, 50(12): 96-103. DOI: 10.16768/j.issn.1004-874X.2023.12.009.
[31] 潘俊峰, 崔克辉, 刘彦卓, 王昕钰, 严博宇, 梁开明. 利用高密度Bin图谱定位水稻叶绿素含量QTL[J]. 广东农业科学, 2022, 49(9): 132-140. DOI: 10.16768/j.issn.1004-874X.2022.09.014. PAN J F, CUI K H, LIU Y Z, WANG X Y, YAN B Y, LIANG K M. Mapping of QTL for chlorophyll content in rice on high-density Bin Map[J]. Guangdong Agricultural Sciences, 2022, 49(9): 132-140. DOI: 10.16768/j.issn.1004-874X.2022.09.014.
[32] 闫晓霞, 朱满山, 王丰, 柳武革, 李金华, 霍兴, 黄永相, 刘迪林. 利用高密度遗传图谱定位水稻耐低氧萌发QTL[J]. 广东农业科学, 2 0 2 3, 5 0 (4) : 1 3 -21. D O I : 10.16 7 6 8 / j. i s s n. 10 0 4 -874X. 2023.04.002. YAN X X, ZHU M S, WANG F, LIU W G, LI J H, HUO X, HUANG Y X, LIU D L. Mapping of QTLs for tolerance of hypoxia germination in rice with a high-density genetic map[J]. Guangdong Agricultural Sciences, 2023, 50(4): 13-21. DOI: 10.16768/j.issn.1004-874X.2023.04.002.
[33] CHEN H, TANG Y Y, LIU J F, TAN L B, JIANG J H, WANG M M, ZHU Z F, SUN X Y, SUN C Q. Emergence of a novel chimeric gene underlying grain number in rice[J]. Genetics, 2017, 205(2): 993-1002. DOI: 10.1534/genetics.116.188201.



 下载:
下载: